分子肿瘤学研究室 Department Introduction
分子肿瘤学研究室成立于2016年,是专注于消化道肿瘤的基因组学、分子生物学和临床转化的开放型研究科室。研究室主任由中国工程院院士詹启敏担任。团队目前有工作人员11人,其中正高2名,副高4名,中级3名,初级2名。
团队研究方向
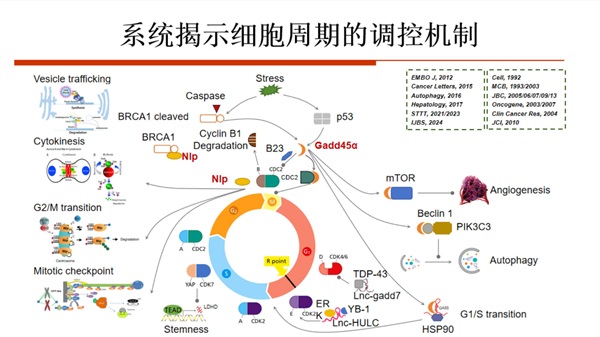
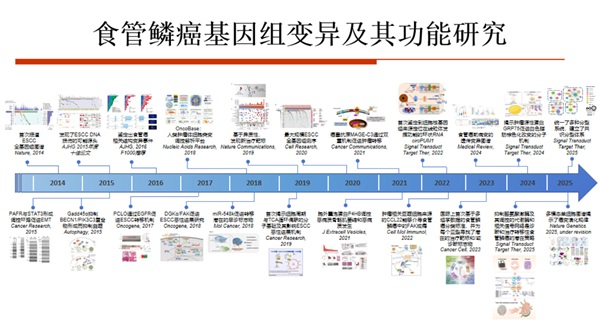
致力于肿瘤分子生物学和转化医学研究,在细胞周期调控、基因组稳定性和细胞癌变机制研究中取得原创性和系统性成果。在国际上率先发现和系统揭示了细胞周期监测点关键蛋白的作用和机制,阐明多个重要细胞周期调控蛋白在细胞癌变和肿瘤诊断与个体化治疗中的作用。从基因组水平和微环境视角全面系统揭示食管鳞癌的遗传变异特征。阐明癌前病变和肿瘤微环境的分子特征和生物学机制,为寻找食管鳞癌诊断的分子标志物提供重要基础。聚焦肿瘤与宿主互作的科学问题,重点研究影响50-80%肿瘤患者的泛组织损害的典型疾病——恶液质。综合应用多组学分析技术和分子生物学技术手段,系统阐明导致恶病质组织损害的肿瘤源性关键分子及其作用机制,为恶病质新的治疗方案提供理论基础和实验依据。
团队近年研究成果

联系我们
咨询电话:010-88121122
地址:北京海淀区阜成路52号(定慧寺)
邮编:100142
友情链接
网站备案

北大肿瘤医院APP

微信服务号

北肿微课堂

微信订阅号
微博






