研究发现肠道微生物预测免疫疗法疗效
英国辛克斯顿维康信托桑格研究所Gunjur等报告,通过深度鸟枪法宏基因组测序技术,对一项Ⅱ期临床队列研究(发现队列)中的106例经组织学证实为晚期实体瘤患者的粪便样本进行了分析,并利用机器监督学习模型证明了,与物种水平的微生物组特征相比,菌株水平的微生物组特征,能更好地预测免疫检查点抑制剂的反应。(Nat Med. 2024, 30: 797-809.)
研究者还在6个相似的队列研究(验证队列)中验证了,菌株水平的微生物组特征的预测能力在不同癌症和不同国家中均具有普适性,不过这一结果只能在发现队列和验证队列使用一致的免疫检查点抑制剂方案时,才能成立。这也意味着,以菌株水平划分的微生物组特征可跨癌种预测免疫治疗疗效,但受免疫治疗方案影响。
为了更好地观察肠道微生物与免疫检查点抑制剂反应之间的关系,研究者纳入CA209-538研究的106例患者的基线粪便样本数据,该研究为前瞻性、多中心的Ⅱ期非随机临床研究,主要评估纳武利尤单抗+伊匹木单抗联合治疗晚期实体瘤的疗效。研究共入组120例晚期实体瘤患者,患者肿瘤类型分为上消化道和胆管癌、神经内分泌肿瘤和罕见妇科肿瘤。
通过对这106个粪便样本进行深度鸟枪法宏基因组测序,研究者得到了一个包含1397个菌株基因组,涵盖904个已知物种,另外还包括34个新菌株(只能分类到属)的数据库。
研究者以监督机器学习的随机森林模型为基础,将临床因素(包括白蛋白水平和中性粒细胞与淋巴细胞比值在内的15个相关指标)和/或微生物组特征(从科、属、种、株水平上的微生物丰度)输入到模型中,分别构建了几个预测患者客观缓解或进展(RvsP)的模型。

结果发现,仅使用临床因素对患者RvsP的预测效果很差(曲线下面积AUC只有0.56),但当使用微生物组特征时,从科到菌株水平的微生物组特征的预测效果依次增强,其中按菌株水平划分的微生物组特征的模型预测性能最好(RvsP的AUC=0.73)。
以上数据表明,肠道微生物特征,尤其是菌株水平的微生物组特征,在预测肿瘤反应方面比更高级别分类或临床特征更有价值。
研究者也试图观察在菌株水平上,具体哪些菌株与治疗反应较好有关,最终研究人员锁定了前22种与治疗反应有关的菌株,其中有20个属于革兰氏阳性菌,18个属于厚壁菌门。与预测反应最相关的菌株们都聚集在瘤胃球菌科的一个分支中,不过遗憾的是,目前只能识别出一个物种F. prausnitzii。
接下来最重要的就是验证在菌株水平下的微生物特征的预测能力能否在不同癌种中复现,于是研究者在6个相似的队列(共涉及五个国家、11个城市的383例癌症患者,同样进行了宏基因组分析,属于验证队列)中进行了验证。结果发现,尽管研究地点不同,但总体上,这些菌株水平的肠道微生物特征在不同癌症(跨癌种)和不同国家中具有普遍有效性(平均AUC=0.65)。
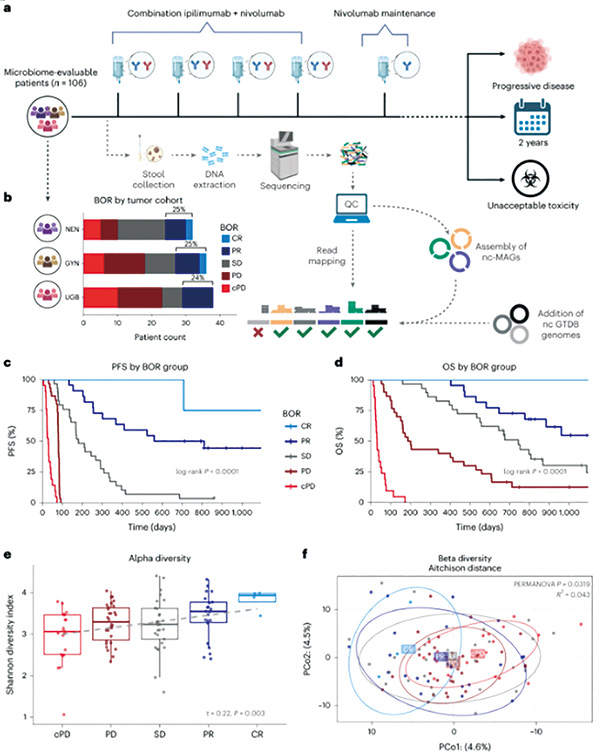
不过,这一结果只有在验证队列与发现队列使用同一种治疗策略时(即联合治疗的CICB方案)才能出现。如果验证队列用的是抗PD-1抗体这样的免疫单药则不具备普适性(平均AUC=0.51)。
研究结果说明,菌株水平的肠道微生物组特征在预测免疫检查点抑制剂治疗反应方面的应用可能需要根据治疗方案进行调整,而不是患者所患癌症的类型。
总而言之,该研究发现,与物种水平的微生物组特征相比,菌株水平的微生物组特征,能更好地预测免疫检查点抑制剂的反应,强调了菌株水平的微生物组作为免疫治疗反应预测标志物的价值。 (编译 张嘉佳)