历时十余年、33种癌症、超11000份样本绘制泛癌图谱
癌症基因组图谱(TCGA)项目最终结果,历时十余年对33种癌症超过11000份样本进行的迄今为止最全面的跨癌基因组分析(Pan-Cancer Atlas),日前以27篇论文集形式在《Cell》系列杂志全盘托出。
除了癌症基因组测序,Pan-Cancer Atlas项目还综合了各种组学数据,包括肿瘤的转录组学、蛋白组学、甲基化组学、临床以及影像学方面的数据,挖掘其中有效信息,绘制出泛癌症图谱,该图谱对肿瘤在人体内如何产生、何处产生、为何产生进行了深入剖析,为未来的临床试验和治疗带来启示。该项目于2005年启动,由美国国立卫生研究院(NIH)资助,耗资3亿美元,由国家人类基因组研究所(NHGRI)和国立癌症研究所(NCI)共同执行。
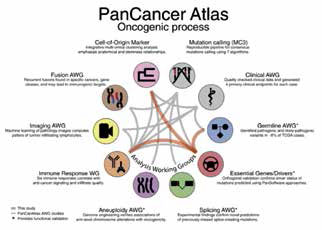
泛癌症图谱根据分子特征对人类癌症类型进行了重新分类,为科研和临床试验设计带来启示。泛癌症图谱数据报告分为三大部分、26篇科研论文,分别发表在《细胞》及其旗下《细胞报告》、《癌细胞》、《细胞系统》、《免疫》等顶尖医学杂志上。每部分由一篇核心论文叙述该部分主要研究成果,其他辅助论文从各角度进行深入的解释。
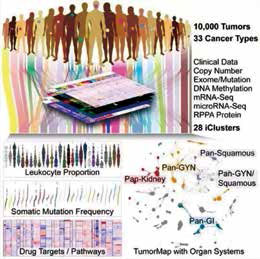
第一篇论文中,研究者采用一种名为iCluster的分子聚类算法,对33种癌症、11286份肿瘤样本的染色体非整倍性、DNA甲基化、mRNA、miRNA和蛋白质数据进行了整合分析。根据分子特征,研究者们将解剖学上的33种癌症重新归类为28种亚型,某一种分子亚型,竟能包括传统解剖学意义上的25种癌症,有助于针对不同类型癌症进行更针对性治疗。
研究表明,源于不用器官的肿瘤可能在分子层面有诸多共性,而来自同一器官的肿瘤可能有截然不同的基因组图谱。该研究中,仅1/3的肿瘤表现出了同质性,其余2/3均有不同程度的异质性。研究者估计,以分子特征作为基准,至少将有1/10的肿瘤患者需要重新分类,可能需要探讨新的治疗方案。这些结果与大量临床研究结合起来,将直接造福肿瘤研究及新药研发。
另一篇研究利用机器学习算法通过对细胞转录组和表观遗传学特征的分析评估癌细胞去分化程度,测定肿瘤细胞的干细胞样特征,鉴定可能的新靶点。研究发现,肿瘤细胞去分化程度与PD-L1水平及免疫微环境相关,且转移性肿瘤细胞的去分化程度更高。
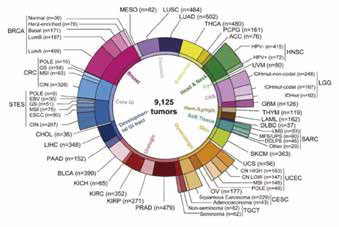
一项研究对9125个肿瘤样本中的突变、拷贝数、mRNA、基因融合和表观遗传数据进行了综合分析,探讨了十种经典通路中基因突变的模式和机制,根据通路变化,研究者把33种癌症分为64种亚型,确定了各通路之间协作和排斥的模式。研究发现,89%的肿瘤至少存在一种通路改变,且根据现有的靶向药物治疗方案,至少57%的肿瘤能得到一种有效的治疗,30%的肿瘤可以进行多靶点治疗,可探讨新的联合治疗方案。

癌症基因图谱的大量数据,使得研究者得以深究癌症中发生的分子变化,对癌症进行更有现实意义的分类,在很多方面取得了始料未及的进步。在研究过程中,对大批量基因数据的处理将计算机方法引入生物科研领域,间接促进了生物信息学的发展,也为其他人类疾病的基因组学研究奠定了基础。
癌症基因组图谱是一项伟大的工程,它的完成离不开贯穿全美和加拿大的20余所科研院校顶尖科学家的通力合作。这使得科研成果达到了个人实验室研究所无法企及的高度,并引导了此后一批大型科研项目的合作模式。
癌症基因组图谱数据库面向科学界和一般群众开放,任何人只要想要,都能接触到这些数据。这一方面令原始数据能得到多角度、多层次的分析,产生新的科研成果;另一方面,科研成果的共享对学界整体发展无疑是极为有利的。
(编译 张亦非)














